- 「ターゲットタンパク研究プログラム(TPRP)」は2007年から5年間実施され2011年度末に一応終了致しましたが、2012年度以後も、ターゲットタンパク研究分野の基本的生命、医学・薬学等への貢献そして食品・環境等の産業利用、ならびに、技術開発研究課題の生産、解析、制御そして情報の各領域から成果がさまざまな形で発表・提供されています。特に、技術研究開発課題の成果は、創薬等支援技術基盤プラットフォーム事業に継承されて、構造生命科学の基盤として機能し始めています。 本ポータルサイトでは、これからも2012年3月までの研究成果の集約を開示し続けるとともに、その後の展開も「構造ギャラリー」と「構造生命科学ニュースウオッチ」でご紹介してまいります。
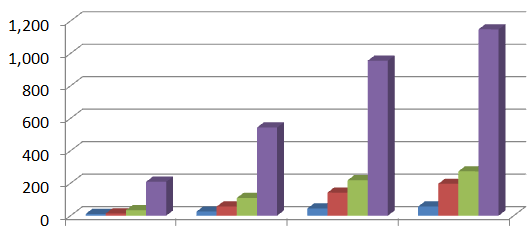
最近公開された構造(2015/08/26 現在)。画像をクリックするとサマリー情報に、IDコードをクリックすると、PDB(Protein Data Bank)データベースで詳細情報にアクセスできます。
 [5AWZ] rhodopsin I |
 [5AX0] rhodopsin I |
 [5AX1] rhodopsin I |
 [4TMB] CmOYE |
本プログラムで取り組まれている膜たんぱく質など464の構造が公開されています(2015/08/26時点)。一覧のダウンロード(Excelファイル)はこちらから。
なお、タンパク3000プロジェクト(2002‐2006年度)で解析されたタンパク質に関する構造ギャラリーは、こちらからご覧になれます。
2013年5月以後も、プログラムの研究成果が発表されてまいりますが、プレスリリースにつきましては、構造生命科学ニュースウオッチの記事としてご紹介してまいります。

 サイト内検索
サイト内検索