- HOME
- 本プログラムについて
- 技術開発「解析」領域
- SAIL法
SAIL法による蛋白質構造解析技術の多様化(H19〜H21)
代表機関:名古屋大学大学院理学研究科
代表研究者:甲斐荘正恒
2010年版パンフレット(背景と成果)の該当ページはこちら
前ページにもあるように、NMRはタンパク質の構造解析手法として広く使われています。X線構造解析ではタンパク質を結晶化させる必要がありますが、NMRは生体内に近い水溶液中のタンパク質を対象とできるのが重要な利点の一つです。
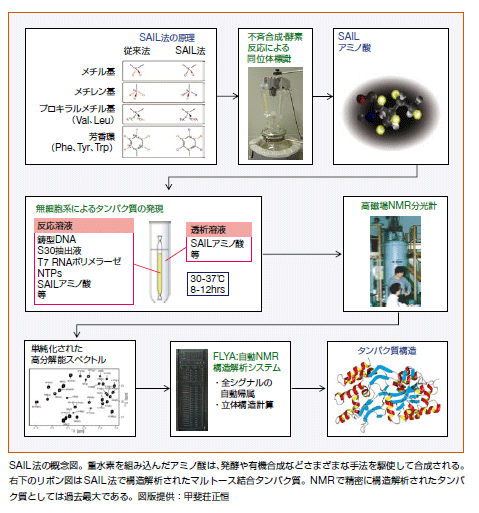
NMRの信号は、原子核同士の位置関係についての情報を与えてくれます。原子数が少なければ、それらの情報からタンパク質の立体構造を解析することは従来の手法でも可能です。しかし、タンパク質が大きくなると、運動が遅くなってNMR信号が幅広くなる上に、信号の数も増えて互いに重なりあうため構造解析は難しくなります。そこで私は、多すぎる情報量をなんとか減らし、しかもより正確な構造を得たいと考え、SAIL法を編み出しました。そのアイディアの鍵は、安定同位元素の利用にあります。
NMRで最もよく利用される水素原子の場合、安定同位元素として通常の水素(H)と原子量が2倍の重水素(D)があります。これらは化学的にはよく似ていますが、NMR信号の共鳴周波数が大きく異なります。天然のタンパク質中の水素原子はほとんどがHなので、それらのうち構造解析に重要な位置だけを残し、残りをDに置換すれば、Hに由来するNMR信号の数は大幅に減り、解析が格段に容易になるはずです。置換する位置を慎重に検討することで、構造決定の精度も上がります。
むろん、タンパク質中の望みの位置のHを、直接Dで置換することはできません。そこで、タンパク質の材料となる20種類のアミノ酸について、さまざまな位置のHをDで置換したものを事前に合成しておきます。炭素や窒素の原子も、必要に応じて同位元素に置換しておきます。こうして得た構造解析に最適な同位体パターンをもつアミノ酸を、目的のタンパク質と同じ順番につなぎます。
このとき、ふつうは大腸菌を使うのですが、大腸菌は増殖する過程で貴重なアミノ酸を消費し、また、苦労して設計した同位体パターンを崩してしまいます。この問題を回避するために、大腸菌からタンパク質合成機能を保った抽出液を調製してタンパク質を合成する「無細胞タンパク質合成系」を採用しました。
このように、目的のタンパク質をNMR測定に最適な状態にすることが、SAIL法の基本思想です。すでに、従来のNMR手法では精密な構造解析が不可能と考えられていた大きなタンパク質においても、SAIL法が有用なことを明らかにしていますが、当課題ではSAIL法の実用性をさらに高め、より広範なタンパク質を解析対象とするための技術基盤を固めます。上記の各プロセスの高度化を図ることでSAIL法にさらなる力を与え、日本発の独創的技術として世界に発信していきたいと思っています。

 サイト内検索
サイト内検索